CH.4.7 _ LightGBM
LightGBM는 XgBoost보다 가볍다.
둘의 예측 성능은 다르지 않으나 걸리는 시간은 짧다.
그 이유는 일반 GBM 계열의 트리 분할 방법과 다르게 Leaf Wise 방식을 사용하기 때문이다.
대부분의 트리 기반 알고리즘은 Level Wise 방식을 사용한다.
Level Wise 방식의 경우 최대한 균형 잡힌 트리를 유지하면서 분할하게 되는데
Leaf Wise 방식의 경우 트리의 균형 보다는 최대 손실 값을 가지는 Leaf node를 지속적으로 분할하게 되면서 비대칭적인 규칙 트리가 생성된다.
균형 잡힌 트리의 경우 과적합 상황에 더 잘 대응하게 되는데, LightGBM의 경우 비대칭적인 트리를 만드는 방식으로 분할 되기 때문에, 자료 수가 1000개 이하인 데이터 세트에서는 과적합이 발생 가능하다.
그러나, 최대 손실 값을 갖는 리프 노드를 지속적으로 분할하기 때문에 결국에 균형 트리 분할 방식보다 예측 오류 손실을 최소화 할 수 있다는 장점이 있다.
# 주요 하이퍼 파라미터
트리의 깊이가 매우 깊어지므로 이런 트리 특성에 맞는 하이퍼 파라미터 설정이 필요하다
- num_iterations (= n_estimator): 반복 수행하는 트리의 개수 지정. 크게 지정할 수록 예측 성능이 높아진다.
*너무 크게 지정시, 과적합으로 성능이 저하 된다.
- learning_rate : num_iterations를 크게 하고, learning_rate를 작게 하면 예측 성능을 향상시킬 수 있다.
- max_depth : 0보다 작은 값을 지정하면 깊이에 제한이 없다.
- min_data_in_leaf (= min_samples_leaf) : 과적합 제어 파라미터
- bagging_fraction : 트리가 커져서 과적합되는 것을 제어하기 위해, 데이터를 샘플링하는 비율을 지정하는 것.
- feature_fraction : 개별 트리를 학습할 때마다 무작위로 선택하는 피쳐의 비율로, 과적합을 제어
- lambda_l2 : L2 regulation 제어를 위한 값으로, 값이 클수록 과적합 감소 효과가 있다.
- lambda_l1 : L1 regulation 제어를 위한 값으로, reg_alpha와 같은 값이다.
# 튜닝 방향
num_leaves의 갯수를 중심으로 min_child_samples, max_depth를 조정해서 모델 복잡도를 감소시키는 방향이다.
즉, 과적합에 대응하는 것.
일반적으로, learning_rate를 작게 하면서, n_estimators를 크게 하는 게 가장 기본적인 방안이다.
그러나 n_estimators를 너무 크게 하는 것은 과적합으로 오히려 성능이 저하될 수 있다.
from lightgbm import LGBMClassifier
import pandas as pd
import numpy as np
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
dataset = load_breast_cancer()
cancer_df = pd.DataFrame(data=dataset.data, columns=dataset.feature_names)
cancer_df['target']= dataset.target
X_features = cancer_df.iloc[:, :-1]
y_label = cancer_df.iloc[:, -1]
X_train, X_test, y_train, y_test=train_test_split(X_features, y_label, test_size=0.2, random_state=156 )
X_tr, X_val, y_tr, y_val= train_test_split(X_train, y_train, test_size=0.1, random_state=156 )
lgbm_wrapper = LGBMClassifier(n_estimators=400, learning_rate=0.05)
evals = [(X_tr, y_tr), (X_val, y_val)]
lgbm_wrapper.fit(X_tr, y_tr, early_stopping_rounds=50, eval_metric="logloss", eval_set=evals, verbose=True)
preds = lgbm_wrapper.predict(X_test)
pred_proba = lgbm_wrapper.predict_proba(X_test)[:, 1]
get_clf_eval(y_test, preds, pred_proba)
from lightgbm import plot_importance
import matplotlib.pyplot as plt
%matplotlib inline
fig, ax = plt.subplots(figsize=(10, 12))
plot_importance(lgbm_wrapper, ax=ax)
plt.savefig('lightgbm_feature_importance.tif', format='tif', dpi=300, bbox_inches='tight')
CH.4.8 _ 베이지안 최적화 기반의 HyperOpt를 이용한 하이퍼 파라미터 튜닝
하이터 파라미터 튜닝을 위해 지금까지 사용한 Grid Search 방식은 하이퍼 파라미터의 수가 많을 경우 최적화 수행 시간이 매우 오래 걸린다는 단점이 있다.
그 대안으로 나온 방법이 '베이지안 최적화 개요'이다.
# 베이지안 최적화 개요
베이지안 확률에 기반을 둔 최적화 기법으로 대체 모델과 획득 함수를 이용한다.
대체 모델은 획득 함수로부터 최적 함수를 예측할 수 있는 입력값을 추천 받은 뒤 이를 기반으로 최적 함수 모델을 개선해 나간다. 획득 함수는 개선된 대체 모델을 기반으로 최적 입력값을 계산한다.
대체 모델은 최적 함수를 추정할 때 다양한 알고리즘을 사용할 수 있는데 일반적으로는 가우시안 프로세스를 적용한다.
그러나 HyperOpt는 트리 파르젠 방식을 사용하게 된다.
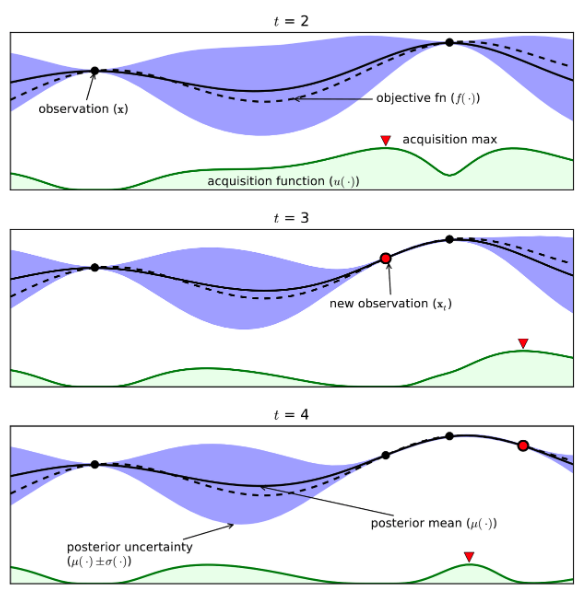
# HyperOpt 이용하기
1. 입력 변수명과 검색 공간을 설정한다
2. 목적 함수 설정
3. 목적 함수의 반환 최솟값을 가지는 최적 입력값을 유추하는
#검색 공간 지정
--> hp.quniform(label, low, high, q) // hp.uniform(label, low, high) // hp.randint(label, upper) // hp.loguniform(label, low, high) // hp.choice(label, options) 같은 함수를 이용할 수 있다.
# 검색 공간 지정
from hyperopt import hp
search_space = {'x': hp.quniform('x', -10, 10, 1), 'y': hp.quniform('y', -15, 15, 1) }# 목적 함수 생성
from hyperopt import STATUS_OK
def objective_func(search_space):
x = search_space['x']
y = search_space['y']
retval = x**2 - 20*y
return retvalfrom hyperopt import fmin, tpe, Trials
import numpy as np
trial_val = Trials()
best_01 = fmin(fn=objective_func, space=search_space, algo=tpe.suggest, max_evals=5
, trials=trial_val, rstate=np.random.default_rng(seed=0))
print('best:', best_01)
trial_val = Trials()
# max_evals를 20회로 늘려서 재테스트
best_02 = fmin(fn=objective_func, space=search_space, algo=tpe.suggest, max_evals=20
, trials=trial_val, rstate=np.random.default_rng(seed=0))
print('best:', best_02)# HyperOpt 이용한 XGBoost 하이퍼 파라미터 최적화
HyperOpt의 경우 입력값과 반환값이 ㅣ모두 실수형이다.
그러나 XGBoost의 경우 정수형 하이퍼 파라미터를 이용해야 하기 때문에 HyperOpt를 이용해 XGBoost 하이퍼 파라미터 최적화를 진행할 때는 정수형으로 형변환을 해줘야 한다.
또한 HyperOpt의 목적 함수는 최솟값을 반환할 수 있도록 최적화해야 하기 때문에 정확도처럼 값이 높을 수록 좋은 성능 지표의 경우, -1을 곱해서 반환해야 한다.
X_train, X_test, y_train, y_test=train_test_split(X_features, y_label, test_size=0.2, random_state=156 )
X_tr, X_val, y_tr, y_val= train_test_split(X_train, y_train, test_size=0.1, random_state=156 )
from hyperopt import hp
xgb_search_space = {'max_depth': hp.quniform('max_depth', 5, 20, 1),
'min_child_weight': hp.quniform('min_child_weight', 1, 2, 1),
'learning_rate': hp.uniform('learning_rate', 0.01, 0.2),
'colsample_bytree': hp.uniform('colsample_bytree', 0.5, 1),
}
from sklearn.model_selection import cross_val_score
from xgboost import XGBClassifier
from hyperopt import STATUS_OK
def objective_func(search_space):
xgb_clf = XGBClassifier(n_estimators=100, max_depth=int(search_space['max_depth']),
min_child_weight=int(search_space['min_child_weight']),
learning_rate=search_space['learning_rate'],
colsample_bytree=search_space['colsample_bytree'],
eval_metric='logloss')
accuracy = cross_val_score(xgb_clf, X_train, y_train, scoring='accuracy', cv=3)
return {'loss':-1 * np.mean(accuracy), 'status': STATUS_OK}
from hyperopt import fmin, tpe, Trials
trial_val = Trials()
best = fmin(fn=objective_func,
space=xgb_search_space,
algo=tpe.suggest,
max_evals=50,
trials=trial_val, rstate=np.random.default_rng(seed=9))
print('best:', best)
print('colsample_bytree:{0}, learning_rate:{1}, max_depth:{2}, min_child_weight:{3}'.format(
round(best['colsample_bytree'], 5), round(best['learning_rate'], 5),
int(best['max_depth']), int(best['min_child_weight'])))
xgb_wrapper = XGBClassifier(n_estimators=400,
learning_rate=round(best['learning_rate'], 5),
max_depth=int(best['max_depth']),
min_child_weight=int(best['min_child_weight']),
colsample_bytree=round(best['colsample_bytree'], 5)
)
evals = [(X_tr, y_tr), (X_val, y_val)]
xgb_wrapper.fit(X_tr, y_tr, early_stopping_rounds=50, eval_metric='logloss',
eval_set=evals, verbose=True)
preds = xgb_wrapper.predict(X_test)
pred_proba = xgb_wrapper.predict_proba(X_test)[:, 1]
get_clf_eval(y_test, preds, pred_proba)
CH.4.9 _ 분류 실습 - 캐글 산탄데르 고객 만족 예측
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import matplotlib
import warnings
warnings.filterwarnings('ignore')
cust_df = pd.read_csv("C:\\ext\\train.csv\\train.csv")
print('dataset shape: ', cust_df.shape)
cust_df.head(3)
print(cust_df['TARGET'].value_counts())
unsatisfied_cnt = cust_df[['TARGET']==1].TARGET.count()
total_cnt = cust_df.TARGET.count()
print('unsatisfied의 비율은 {0:.2f}'.format((unsatisfied_cnt/total_cnt)))
cust_df.describe()
cust_df['var3'].replace(-999999, 2, inplace=True)
cust_df.drop('ID', axis=1, inplace=True)
X_features = cust_df.iloc[:, :-1]
y_labels = cust_df.iloc[:, -1]
print('피처 데이터 shape: {0}'.format(X_features.shape))
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X_features, y_labels, test_size=0.2, random_state=0)
train_cnt = y_train.count()
test_cnt = y_test.count()
print('학습 세트 Shape : {0}, 테스트 세트 Shape : {1}'.format(X_train.shape, X_test.shape))
print('학습 세트 레이블 값 분포 비율')
print(y_train.value_counts()/train_cnt)
print('\n 테스트 세트 레이블 값 분포 비율')
print(y_test.value_counts()/test_cnt)
X_tr, X_val, y_tr, y_val = train_test_split(X_train, y_train, test_size=0.3, random_state=0)
from xgboost import XGBClassifier
from sklearn.metrics import roc_auc_score
xgb_clf = XGBClassifier(n_estimators=500, learning_rate=0.05, random_state=156)
xgb_clf.fit(X_tr, y_tr, early_stopping_rounds=100, eval_metric="auc", eval_set=[(X_tr, y_tr), (X_val, y_val)])
xgb_roc_score = roc_auc_score(y_test, xgb_clf.predict_proba(X_test)[:, 1])
print('ROC AUC: {0:.4f}'.format(xgb_roc_score))
from hyperopt import hp
xgb_search_space = {'max_depth':hp.quniform('max_depth', 5, 15, 1),
'min_child_weight':hp.quniform('min_child_weight', 1, 6, 1),
'colsample_bytree':hp.uniform('colsample_bytree', 0.5, 0.95),
'learning_rate':hp.uniform('learning_rate', 0.01, 0.2)}
from sklearn.model_selection import KFold
from sklearn.metrics import roc_auc_score
def objective_func(search_space):
xgb_clf= XGBClassifier(n_estimator=100, max_depth=int(search_space['max_depth']),
min_child_weight=int(search_space['min_child_weight']),
colsample_bytree=search_space['colsample_bytree'],
learning_rate=search_space['learning_rate'])
roc_auc_list = []
kf = KFold(n_splits=3)
for tr_index, val_index in kf.split(X_train):
X_tr, y_tr = X_train.iloc[tr_index], y_train.iloc[tr_index]
X_val, y_val = X_train.iloc[val_index], y_train.iloc[val_index]
xgb_clf.fit(X_tr, y_tr, early_stopping_rounds=30, eval_metric='auc',
eval_set=[(X_tr, y_tr), (X_val, y_val)])
score = roc_auc_score(y_val, xgb_clf.predict_proba(X_val)[:,1])
roc_auc_list.append(score)
return -1*np.mean(roc_auc_list)
from hyperopt import fmin, tpe, Trials
trials = Trials()
best = fmin(fn=objective_func, space=xgb_search_space, algo=tpe.suggest, max_evals=50, trials=trials, rstate=np.random.default_rng(seed=30))
print('best', best)
xgb_clf = XGBClassifier(n_estimator=500, max_depth=int(best['max_depth']),
min_child_weight=int(best['min_child_weight']),
colsample_bytree=round(best['colsample_bytree']),
learning_rate=rount(best['learning_rate'],5))
xgb_clf.fit(X_tr, y_tr, early_stopping_rounds=100, eval_metric="auc", eval_set=[(X_tr, y_tr), (X_val, y_val)])
xgb_roc_score = roc_auc_score(y_test, xgb_clf.predict_proba(X_test)[:, 1])
CH.4.10 _ 분류 실습 - 캐글 신용카드 사기 검출
#언더 샘플링과 오버 샘플링
_레이블이 불균형한 분포를 가진 데이터를 학습 시킬 경우, 예측 성능의 문제가 발생할 수 있다.
이상 레이블을 가지는 데이터 건수가 정상 레이블을 가진 데이터 건수에 비해 너무 적을 때 발생
>> 이상 레이블을 가지는 데이터 건수가 너무 적어 다양한 유형을 학습하지 못하지만,
정상 레이블을 가지느느 데이터 건수가 매우 많아 정상 레이블로 치우친 학습을 수행해, 제대로 된 이상 데이터 검출이 어려워지기 쉽다.
--> 이런 내용을 극복하는 방법이 '언더 샘플링'과 '오버 샘플링'이다
1. 언더 샘플링
많은 데이터 세트를 적은 데이터 세트 수준으로 감소 시키는 방법
--> 정상 레이블을 가진 데이터 세트를 줄여 버리는 방식
>> 제대로 된 학습을 수행할 수 없을 수도 있으니 사용에 유의하기
2. 오버 샘플링
적은 데이터 세트를 증식시켜 학습을 위한 데이터를 확보하는 방법
--> 단순히 동일한 데이터를 증식시키는 방법이 아니라, 원본 데이터의 피처 값을 변경시켜 증식시킨다.
Ex) SMOTE 방법
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import warnings
warnings.filerwarnings('ignore')
%matplotlib inline
card_df = pd.read_csv("C:\\ext\\creditcard.csv\\creditcard.csv")
card_df.head(3)
from sklearn.model_selection import train_test_split
def get_preprocessed_df(df=None):
df_copy = df.copy()
df_copy.drop('Time', axis=1, inplace=True)
return df_copy
def get_train_test_dataset(df=None):
df_copy=get_preprocessed_df(df)
X_features = df_copy.iloc[:, :-1]
y_target = df_copy.iloc[:, -1]
X_train, X_test, y_train, y_test = \
train_test_split(X_features, y_target, test_size=0.3, random_state=0, stratify=y_target)
return X_train, X_test, y_train, y_test
X_train, X_test, y_train, y_test = get_train_test_dataset(card_df)
from sklearn.linear_model import LogisticRegression
lr_clf = LogisticRegression(max_iter=1000)
lr_clf.filt(X_train, y_train)
lr_pred = lr_clf.predict(X_test)
lr_pred_proba = lr_clf.predict_proba(X_test)[:,1]
get_clf_eval(y_test, lr_pred, lr_pred_proba)
def get_model_train_eval(model, ftr_train=None, ftr_test=None, tgt_train=None, tgt_test=None):
model.fit(ftr_train, tgt_train)
pred = model.predict(ftr_test)
pred_proba = model.predict_proba(ftr_test)[:,1]
get_clf_eval(tgt_test, pred, pred_proba)
from lightgbm import LGBMClassifier
lgbm_clf = LGBMClassifier(n_estimators=1000, num_leaves=64, n_jobs=-1, boost_from_average=False)
get_model_train_eval(lgbm_clf, ftr_train=X_train, ftr_test=X_test, tgt_train=y_train, tgt_test=y_test)
#데이터 분포도 변환 후 모델 학습/예측/평가
1. Amount를 표준 정규 분포 형태로 변환 후 로지스틱 회귀의 예측 성능 측정
2. 로그 변환 수행 : 데이터 분포도가 심하게 왜곡됐을 경우 적용하는 중요 기법
from sklearn.preprocessing import StandardScaler
def get_preprocessed_df(df=None):
df_copy = df.copy()
scaler = StandardScaler()
amount_n = scaler.fit_transform(df_copy['Amount'].values.reshape(-1,1))
df_copy.insert(0, 'Amount_Scaled', amount_n)
df_copy.drop(['Time', 'Amount'], axis=1, inplace=True)
return df_copy
X_train, X_test, y_train, y_test = get_train_test_dataset(card_df)
def get_preprocessed_df(df=None):
df_copy = df.copy()
amount_n = np.log1p(df_copy['Amount']
df_copy.insert(0, 'Amount_Scaled', amount_n)
df_copy.drop(['Time', 'Amount'], axis=1, inplace=True)
return df_copy
X_train, X_test, y_train, y_test = get_train_test_dataset(card_df)
#이상치 데이터 제거 후 모델 학습/예측/평가
이상치 데이터는 전체 데이터의 패턴에서 벗어난 이상 값을 가진 데이터다.
이상치로 인해 머신러닝 모델의 성능에 영향을 받는 경우가 발생하기 쉽다.
>> 이상치 데이터를 제거하고, 제거한 후 모델의 성능을 평가
#히트맵
import seaborn as sns
plt.figure(figsize=(9,9))
corr = card_df.corr()
sns.heatmap(corr, cmap='RdBu')
import numpy as np
def get_outlier(df=None, column=None, weight=1.5):
fraud = df[df['Class']==1][column]
quantile_25 = np.percentile(fraud.values, 25)
quantile_75 = np.percentile(fraud.values, 75)
iqr = quantile_75 - quantile_25
iqr_weigth = iqr*weight
lowest_val = quantile_25 - iqr_weight
highest_val = quantile_75 + iqur_weight
outlier_index = fraud[(fraud<lowest_val) | (fraud > highest_val)].index
return outlier_index
outlier_index = get_outlier(df=card_df, column='V14', weight=1.5)
def get_preprocessed_df(df=None):
df_copy = df.copy()
amount_n = np.log1p(df_copy['Amount'])
df_copy.insert(0, 'Amount_Scaled', amount_n)
df_copy.drop(['Time', 'Amount'], axis=1, inplace=True)
outlier_index = get_outlier(df=df_copy, column='V14', weight=1.5)
df_copy.drop(outlier_index, axis=0, inplace=True)
return df_copy
X_train, X_test, y_train, y_test = get_train_test_dataset(card_df)
get_model_train_eval(lr_clf, ftr_train=X_train, ftr_test=X_test, tgt_train=y_train, tgt_test=y_test)
get_model_train_eval(lgbm_clf, ftr_train=X_train, ftr_test=X_test, tgt_train=y_train, tgt_test=y_test)
#SMOTE 오버 샘플링 적용 후 모델 학습/예측/평가
SMOTE 기법으로 오버 샘플링을 적용한 후 로지스틱 회귀와 LightGBM 모델의 예측 성능 평가
from imblearn.over_sampling import SMOTE
smote = SMOTE(random_state=0)
X_train_over, y_train_over = smote.fit_resample(X_train, y_train)
lr_clf = LogisticRegression(max_iter=1000)
get_model_train_eval(lr_clf, ftr_train=X_train_over, ftr_test=X_test, tgt_train=y_train_over, tgt_test = y_test)
precision_recall_curve_plot(y_test, lr_clf.predict_proba(X_test)[:,1])
lgbm_clf = LGBMClassifier(n_estimators=1000, num_leaves=64, n_jobs=-1, boost_from_average=False)
get_model_train_eval(lgbm_clf, ftr_train=X_train_over, ftr_test=X_test, tgt_train=y_train_over, tgt_test=y_test)
CH.4.11 _ 스태깅 앙상블
스태킹은 개별적인 여러 알고리즘을 서로 결합해 예측 결과를 도출한다. --> 배깅, 부스팅과의 공통점
개별 알고리즘으로 예측한 데이터를 기반으로 다시 예측을 수행 --> 가장 큰 차이점
<-> 메타 모델
스태킹 모델 <- 개별적인 기반 모델 & 최종 메타 모델 2가지 필요
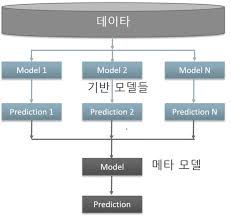
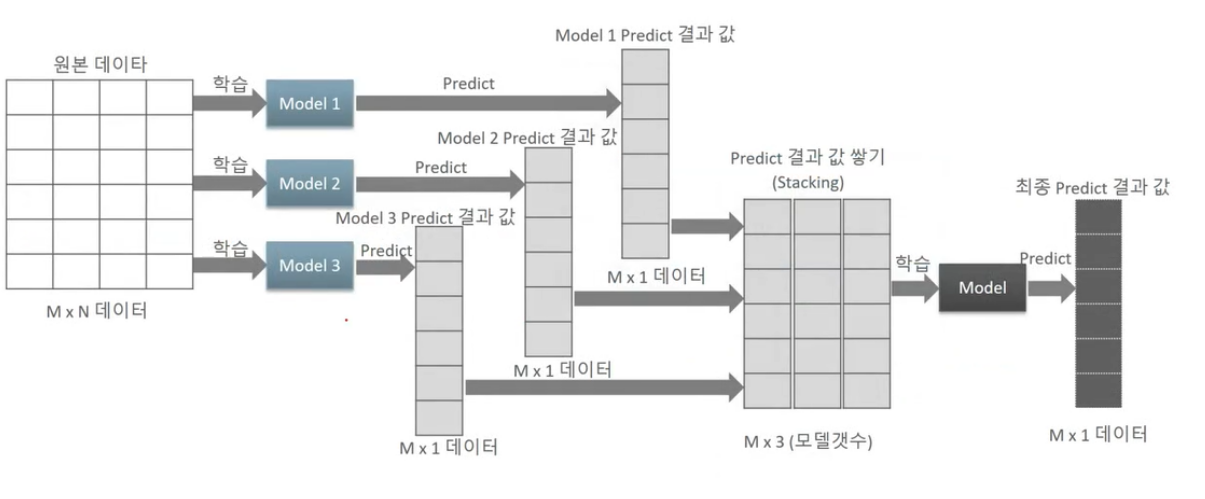
# 기본 스태킹 모델
import numpy as np
from sklearn.neighbors imort KNeighborsClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.ensemble import AdaBoodstClassifier
from sklearn.tree import DecisionTreeClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
cancer_data = load_breast_cancer()
X_data=cancer_data.data
y_label = cancer_data.target
X_train, X_test, y_train, y_test = train_test_split(X_data,y_label,test_size=0.2, random_state=0)
knn_clf = KNeighborsClassifier(n_neighbors=4)
rf_clf = RandomForestClassifier(n_estimators=100, random_state=0)
dt_clf= DecisionTreeClassifier()
ada_clf = AdaBoostClassifier(n_estimators=100)
lr_final = LogisticRegression()
knn_clf.fit(X_train,y_train)
rf_clf.fit(X_train,y_train)
dt_clf.fit(X_train,y_train)
ada_clf.fit(X_train,y_train)
knn_pred = knn_clf.predict(X_test)
rf_pred = rf_clf.predict(X_test)
dt_pred = dt_clf.predict(X_test)
ada_pred = ada_clf.predict(X_test)
pred = np.array([knn_pred, rf_pred, dt_pred, ada_pred])
pred = np.transpose(pred)
lr_final.fit(pred, y_test)
final = lr_final.predict(pred)
# CV 세트 기반의 스태킹
과적합을 개서하기 위해 교차 검증 기반으로 예측된 결과 데이터 세트를 이용한다.
STEP1 : 각 모델별로 원본 학습/테스트 데이터를 예측한 결과 값을 기반으로 메타 모델을 위한 학습용/테스트용 데이터를 생성
STEP2 :
- 스텝 1에서 개별 모델들이 생성한 학습용 데이터를 스태킹 형태로 합쳐, 메타 모델이 학습할 최종 학습용 데이터 세트 생성
- 각 모델의 테스트 데이터를 스태킹 형태로 합쳐, 최종 테스트 데이터 세트 생성
- 메타 모델은 원본 학습 데이터 세트, 스태킹 학습 데이터 세트를 기반으로 학습 & 최종 테스트 데이터 세트 예측 --> 원본 테스트 데이터의 레이블 기반으로 평가 진행
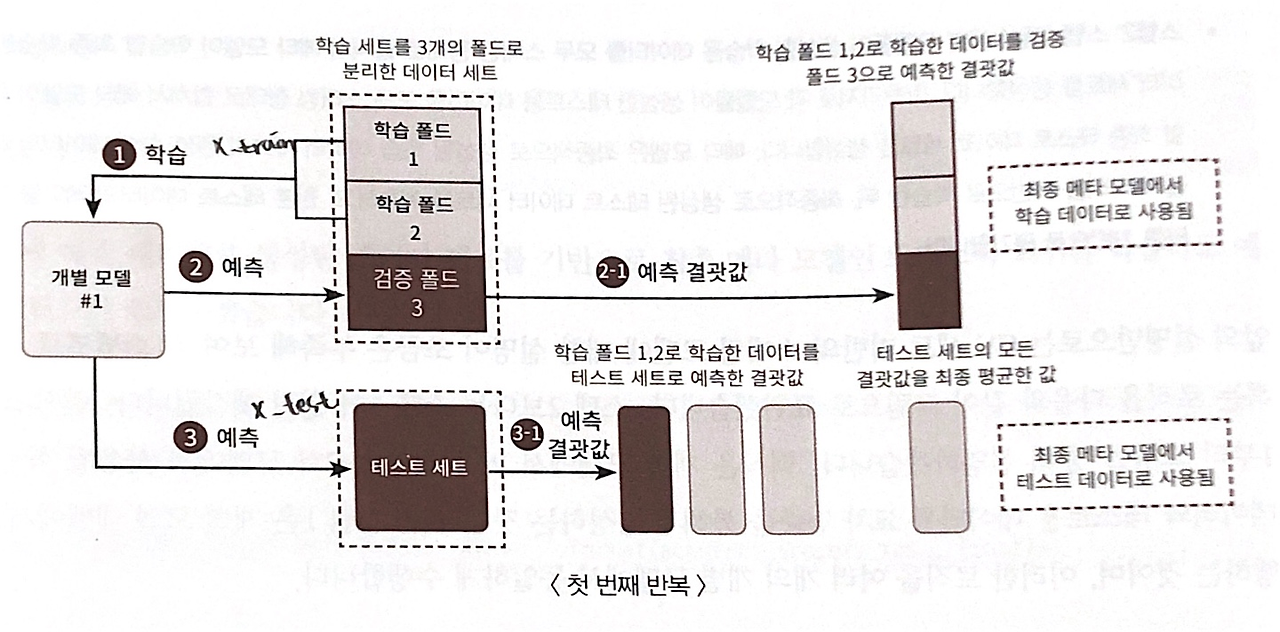
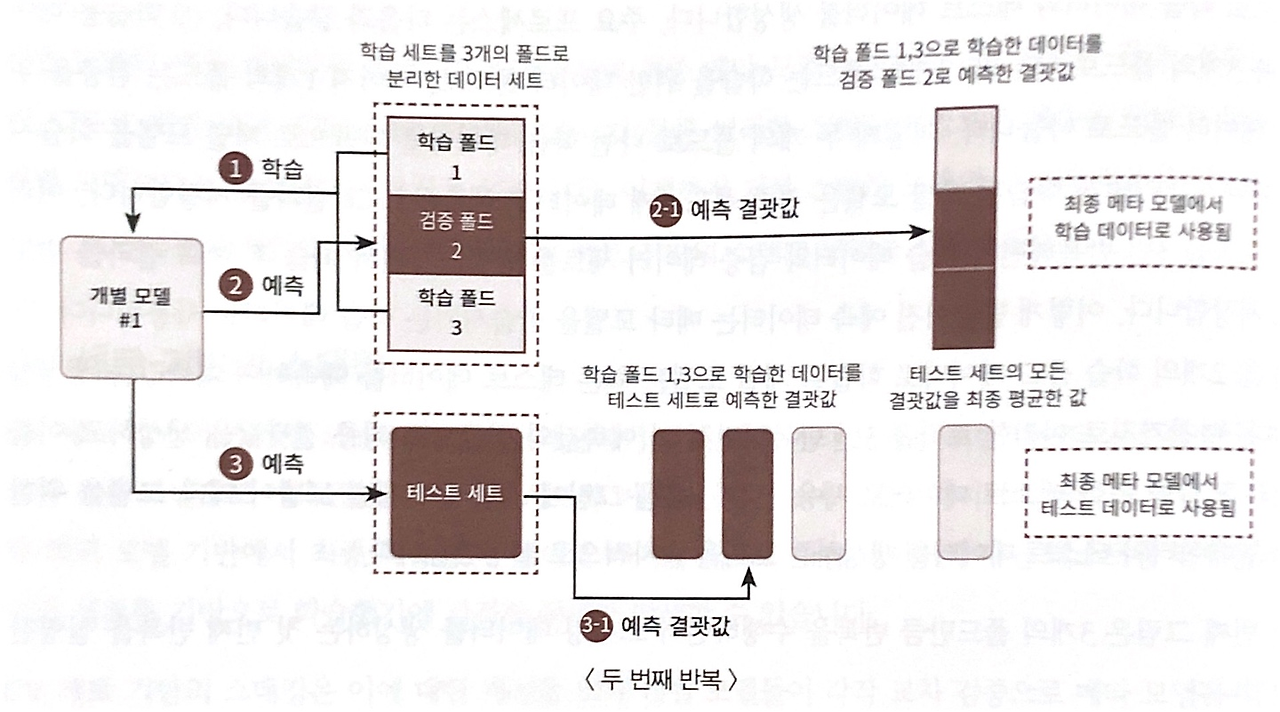
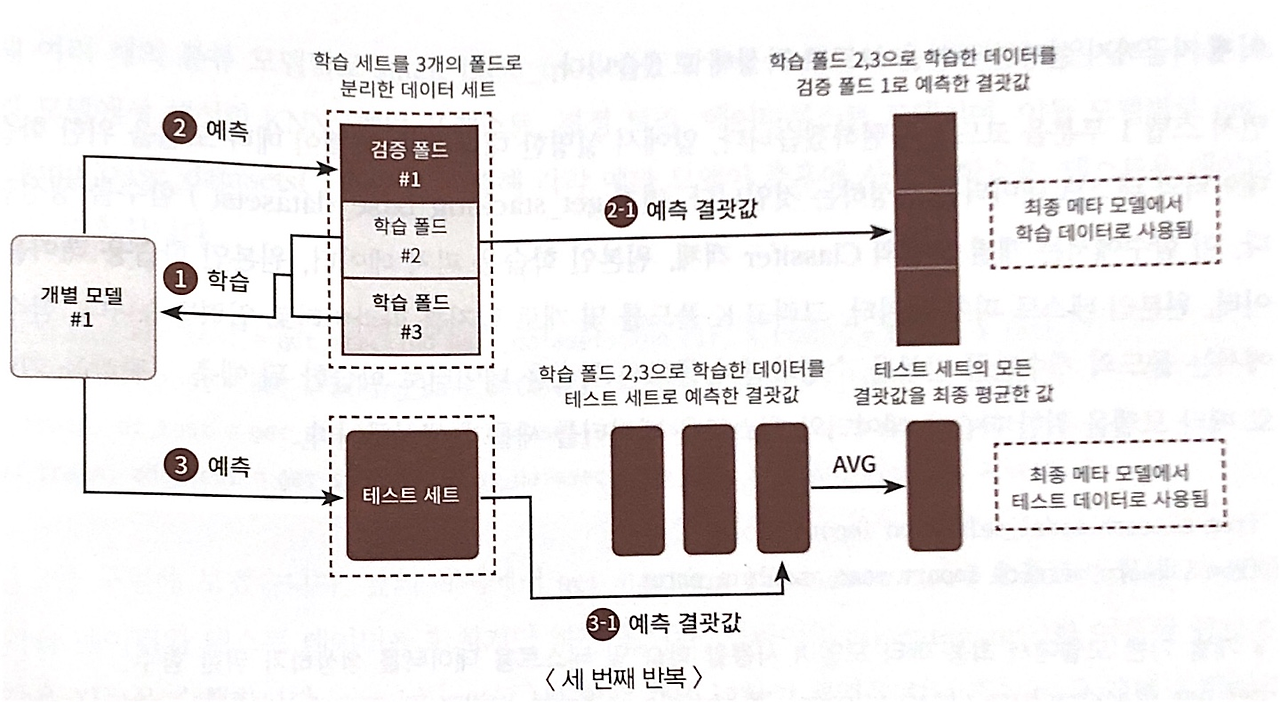
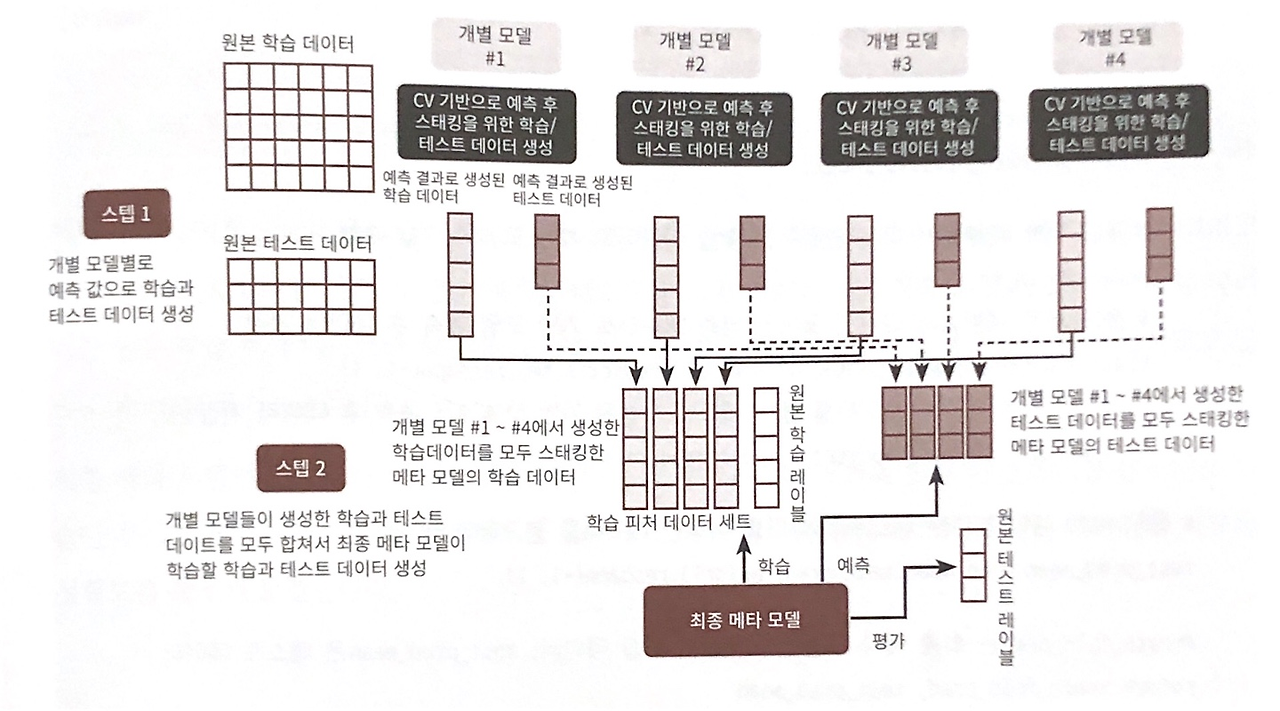
from sklearn.model_selection import KFold
from sklearn.metrics import mean_absolute_error
def get_stacking_base_datasets(model, X_train_n, y_train_n, X_test_n, n_folds ):
kf = KFold(n_splits=n_folds, shuffle=False, random_state=0)
train_fold_pred = np.zeros((X_train_n.shape[0] ,1 ))
test_pred = np.zeros((X_test_n.shape[0],n_folds))
print(model.__class__.__name__ , ' model 시작 ')
for folder_counter , (train_index, valid_index) in enumerate(kf.split(X_train_n)):
print('\t 폴드 세트: ',folder_counter,' 시작 ')
X_tr = X_train_n[train_index]
y_tr = y_train_n[train_index]
X_te = X_train_n[valid_index]
model.fit(X_tr , y_tr)
train_fold_pred[valid_index, :] = model.predict(X_te).reshape(-1,1)
test_pred[:, folder_counter] = model.predict(X_test_n)
test_pred_mean = np.mean(test_pred, axis=1).reshape(-1,1)
#train_fold_pred는 최종 메타 모델이 사용하는 학습 데이터, test_pred_mean은 테스트 데이터
return train_fold_pred , test_pred_mean
knn_train, knn_test = get_stacking_base_datasets(knn_clf, X_train, y_train, X_test, 7)
rf_train, rf_test = get_stacking_base_datasets(rf_clf, X_train, y_train, X_test, 7)
dt_train, dt_test = get_stacking_base_datasets(dt_clf, X_train, y_train, X_test, 7)
ada_train, ada_test = get_stacking_base_datasets(ada_clf, X_train, y_train, X_test, 7)
Stack_final_X_train = np.concatenate((knn_train, rf_train, dt_train, ada_train), axis=1)
Stack_final_X_test = np.concatenate((knn_test, rf_test, dt_test, ada_test), axis=1)
print('원본 학습 피처 데이터 Shape:',X_train.shape, '원본 테스트 피처 Shape:',X_test.shape)
print('스태킹 학습 피처 데이터 Shape:', Stack_final_X_train.shape,
'스태킹 테스트 피처 데이터 Shape:',Stack_final_X_test.shape)
lr_final.fit(Stack_final_X_train, y_train)
stack_final = lr_final.predict(Stack_final_X_test)
print('최종 메타 모델의 예측 정확도: {0:.4f}'.format(accuracy_score(y_test, stack_final)))
CH.4.12 _ 정리
앙상블 기법은 결정 트리 기반의 다수의 약한 학습기를 결합해 변동성을 줄여 예측 오류를 줄이고 성능을 개선한다.
결정트리 알고리즘 - 정보의 균일도에 기반한 규칙 트리 생성 후 예측 수행 --> 직관적이지만, 과적합 발생 쉬움
앙상블 기법 - 배깅(랜덤 포레스트), 부스팅(GBM, SGboost, LightGBM)
스태킹 모델 - 여러 개의 개별 모델이 생성한 예측 데이터를 최종 메타 모델이 학습할 별도의 학습 데이터 세트와 예측할 테스트 데이터 세트를 재생성하는 기법 --> 메타 모델이 사용할 학습, 예측 데이터 세트를 개별 모델의 예측 값을 스태킹 형태로 결합해 생성한다는게 특징적이다.
'Self-Taught > Machine Learning' 카테고리의 다른 글
| 파이썬 머신러닝 완벽 가이드 _CH.6 (0) | 2024.11.23 |
|---|---|
| 파이썬 머신러닝 완벽 가이드 _CH.5 (0) | 2024.11.16 |
| 파이썬 머신러닝 완벽 가이드 _ CH.4.1~4.6 (0) | 2024.11.02 |
| 파이썬 머신 러닝 완벽 가이드 _ CH.3 (2) | 2024.10.12 |
| 파이썬 머신러닝 완벽가이드 _ CH.2_(1) (1) | 2024.10.05 |


