철솜_STUDY
머신러닝 완벽 가이드 _ CH.7 : 군집화 본문
CH.6.1 _K-평균 알고리즘 이해
- K-평균 : 군집화에서 가장 일반적으로 사용하는 알고리즘
- 군집 중심점(centroid)라는 특정한 임의의 지점을 선택해 해당 중심에 가장 가까운 포인트들을 선택하는 군집화 기법
- K 평균의 동작 원리

- 군집화의 기준이 되는 중심을 구성하려는 군집화 개수만큼 임의의 위치에 놓음. 전체 데이터를 2개로 군집화한다면, 2개의 중심을 임의의 위치에 가져다 놓는 것
- 각 데이터는 가장 가까운 곳에 속한 중심점에 소속됨.
- 소속이 결정되면 군집 중심점을 소속된 데이터의 평균 중심으로 이동시킴.
- 중심점이 이동했기에 각 데이터는 기존에 속한 중심점보다 더 가까운 중심점이 존재한다면, 해당 중심점으로 다시 소속을 변경함.
- 다시 중심을 소속된 데이터의 평균 중심으로 이동함. 위 그림에서는 데이터 C가 중심 소속이 변경되면서 두 개의 중심이 모두 이동
- 중심점을 이동했는데 데이터의 중심점 소속 변경이 없으면, 군집화 종료. 아니라면, 다시 4번 과정부터 반복
- K-평균의 장점
- 일반적인 군집화에서 가장 많이 사용하는 알고리즘
- 알고리즘이 쉽고 간결
- K-평균의 단점
- 거리 기반 알고리즘으로 속성의 개수가 매우 많은 경우 군집화 정확도가 떨어짐. (PCA필요 가능성 있음)
- 반복을 수행하는 경우, 반복 횟수가 많을 경우, 수행시간이 매우 느려짐
- 몇개의 군집을 선택해야 할지 가이드하기가 어려움.
사이킷런 KMeans 클래스 소개
- 중요 파라미터
- n_clusters : 군집화할 개수. 즉, 군집 중심점의 개수
- init : 초기에 군집 중심점의 좌표를 설정한 방식. 일반적으로 k-means++ 방식으로 최초설정
- max_iter : 최대 반복 횟수. 이 횟수 이전에 모든 데이터의 중심점 이동이 없으면 종료
- KMeans 주요 속성 정보
- labels_ : 각 데이터 포인트가 속한 군집 중심점 레이블
- clustercenters : 각 군집 중심점 좌표 (shape : [군집 개수, 피처 개수]) → 이를 이용해 군집 중심점 좌표가 어디인지 시각화 가능
군집화 알고리즘 테스트를 위한 데이터 생성
- 군집화용 데이터 생성기
- make_blobs() : 개별 군집의 중심점과 표준 편차 제어 기능 추가
- n_samples : 생성할 총 데이터의 개수
- n_features : 데이터의 피처 개수, 시각화를 목표로 할 경우, 2개로 설정해 보통 첫번째 피처 좌표는 x좌표, 두번째 피처 좌표는 y좌표 상에서 표현.
- centers : int값, int의 경우 군집의 개수, ndarray 형태인 경우 개별 군집 중심점의 좌표 나타냄
- cluster_std : 생성될 군집 데이터의 표준 편차. 군집별로 서로 다른 표준 편차를 가진 데이터 세트를 만들때 사용.
- make_classification() : 노이즈를 포함한 데이터를 만드는데 유용하게 사용
- make_blobs() : 개별 군집의 중심점과 표준 편차 제어 기능 추가
from sklearn.preprocessing import scale
from sklearn.datasets import load_iris
from sklearn.cluster import KMeans
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
%matplotlib inline
iris = load_iris()
irisDF = pd.DataFrame(data=iris.data, columns=['sepal_length','sepal_width','petal_length','petal_width'])
irisDF.head(3)kmeans = KMeans(n_clusters=3, init='k-means++', max_iter=300,random_state=0).fit(irisDF)
print(kmeans.labels_)
irisDF['target'] = iris.target
irisDF['cluster']=kmeans.labels_
iris_result = irisDF.groupby(['target','cluster'])['sepal_length'].count()
print(iris_result)from sklearn.decomposition import PCA
pca = PCA(n_components=2)
pca_transformed = pca.fit_transform(iris.data)
irisDF['pca_x'] = pca_transformed[:,0]
irisDF['pca_y'] = pca_transformed[:,1]
irisDF.head(3)
# cluster 값이 0, 1, 2 인 경우 추출
marker0_ind = irisDF[irisDF['cluster']==0].index
marker1_ind = irisDF[irisDF['cluster']==1].index
marker2_ind = irisDF[irisDF['cluster']==2].index
# cluster값 0, 1, 2 >> marker 별도로
plt.scatter(x=irisDF.loc[marker0_ind,'pca_x'], y=irisDF.loc[marker0_ind,'pca_y'], marker='o')
plt.scatter(x=irisDF.loc[marker1_ind,'pca_x'], y=irisDF.loc[marker1_ind,'pca_y'], marker='s')
plt.scatter(x=irisDF.loc[marker2_ind,'pca_x'], y=irisDF.loc[marker2_ind,'pca_y'], marker='^')
plt.xlabel('PCA 1')
plt.ylabel('PCA 2')
plt.title('3 Clusters Visualization by 2 PCA Components')
plt.show()import numpy as np
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
from sklearn.datasets import make_blobs
%matplotlib inline
X, y = make_blobs(n_samples=200, n_features=2, centers=3, cluster_std=0.8, random_state=0)
print(X.shape, y.shape)
unique, counts = np.unique(y, return_counts=True)
print(unique,counts)
import pandas as pd
clusterDF = pd.DataFrame(data=X, columns=['ftr1', 'ftr2'])
clusterDF['target'] = y
clusterDF.head(3)
target_list = np.unique(y)
markers=['o', 's', '^', 'P','D','H','x']
# target==0, target==1, target==2 로 scatter plot을 marker별로 생성.
for target in target_list:
target_cluster = clusterDF[clusterDF['target']==target]
plt.scatter(x=target_cluster['ftr1'], y=target_cluster['ftr2'], edgecolor='k', marker=markers[target] )
plt.show()
## KMeans 객체를 이용하여 X 데이터를 K-Means 클러스터링 수행
kmeans = KMeans(n_clusters=3, init='k-means++', max_iter=200, random_state=0)
cluster_labels = kmeans.fit_predict(X)
clusterDF['kmeans_label'] = cluster_labels
centers = kmeans.cluster_centers_
unique_labels = np.unique(cluster_labels)
markers=['o', 's', '^', 'P','D','H','x']
for label in unique_labels:
label_cluster = clusterDF[clusterDF['kmeans_label']==label]
center_x_y = centers[label]
plt.scatter(x=label_cluster['ftr1'], y=label_cluster['ftr2'], edgecolor='k',
marker=markers[label] )
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=200, color='white',
alpha=0.9, edgecolor='k', marker=markers[label])
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=70, color='k', edgecolor='k',
marker='$%d$' % label)
plt.show()
print(clusterDF.groupby('target')['kmeans_label'].value_counts())CH.7.2 _ 군집 평가
- 군집화가 효율적으로 잘 되었는지 성능평가 → 비지도학습의 특성상 어떠한 지표라도 정확하게 성능을 평가하기는 어려움.
- 군집화 성능 평가의 대표적인 방법으로 실루엣 분석 이용
실루엣 분석의 개요
- 실루엣 분석 : 각 군집 간의 거리가 얼마나 효율적으로 분리되어 있는지 나타냄
>> 효율적으로 잘 분리되었다 : 다른 군집과의 거리는 떨어져 있고, 동일 군집끼리의 데이터는 서로 가깝게 잘 뭉쳐 있음. → 잘 될 수록, 개별 군집은 비슷한 정도의 여유 공간을 가지고 떨어져 있을 것임.
- 실루엣 분석은 실루엣 계수를 기반으로 함
- 실루엣 계수 : 개별 데이터가 가지는 군집화 지표, 해당 데이터가 같은 군집 내의 데이터와 얼마나 가깝게 군집화돼있고, 다른 군집에 있는 데이터와는 얼마나 멀리 분리되어 있는지 나타내는 지표

- 실루엣 계수 식
- : 해당 데이터 포인트와 같은 군집 내에 있는 다른 데이터 포인트와의 거리를 평균한 값
- : 해당 포인트가 속하지 않은 군집 중 가장 가까운 군집과의 평균 거리
- : 두 군집간의 거리가 얼마나 떨어져 있는지
→ 실루엣 계수는 -1에서 1사이의 값을 가지며, 1로 가까워질수록 근처의 군집과 더 멀리 떨어져 있다는 것, 0에 가까울수록 근처의 군집과 가까워진다는 것. -값은 아예 다른 군집에 데이터 포인트가 할당되었음.
사이킷 런에서의 메서드
sklearn.metrics.silhoutte_samples(X, labels, metric=’euclidean’,**kwds)→ 인자로 X feature데이터 세트와 각 피처 데이터 세트가 속한 군집 레이블 값인 label 데이터를 입력해주면 각 데이터 포인트의 실루엣 계수 반환
sklearn.metrics.silhoutte_score(X, labels, metric=’euclidean’, sample_size=None,**kwds)→ 인자로 X feature 데이터 세트와 각 피처 데이터 세트가 속한 군집 레이블 값인 labels 데이터를 입력해주면 전체 데이터의 실루엣 계수 값을 평균해 반환. =np.mean(silhouette_samples())
→ 이 값이 높을수록 군집화가 어느정도 잘 됐다고 판단할 수 있음. 하지만 이 값이 높다고 해서 군집화가 잘 됐다고 판단할 수 없음.
- 좋은 군집화의 조건
- 전체 실루엣 계수의 평균 값이 0~1 사이의 값을 가지며, 1에 가까울수록 좋음
- 전체 실루엣 계수의 평균과 더불어 개별 군집의 평균값의 편차가 크지 않아야 함. 즉, 개별 군집의 실루엣 계수 평균값이 전체 실루엣 계수의 평균값에서 크게 벗어나지 않는 것이 중요
군집별 평균 실루엣 계수의 시각화를 통한 군집 개수 최적화 방법
- 전체 데이터의 평균 실루엣 계수 값이 높다고 해서 반드시 최적의 군집 개수로 군집화가 잘 됐다고 볼 수 없음.
- 특정 군집 내의 실루엣 계수 값만 너무 높고, 다른 군집은 내부 데이터끼리 거리가 너무 멀리 떨어져 있어 실루엣 계수 값이 낮아져도 평균적으로 높은 값을 가질 수 있음.
→ 개별 군집별로 적당히 분리된 거리를 유지하면서도 군집 내의 데이터가 서로 뭉쳐있는 경우에 K-평군의 적절한 군집 개수가 설정되었다고 판단할 수 있음.
from sklearn.preprocessing import scale
from sklearn.datasets import load_iris
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_samples, silhouette_score
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
%matplotlib inline
iris = load_iris()
feature_names = ['sepal_length','sepal_width','petal_length','petal_width']
irisDF = pd.DataFrame(data=iris.data, columns=feature_names)
kmeans = KMeans(n_clusters=3, init='k-means++', max_iter=300,random_state=0).fit(irisDF)
irisDF['cluster'] = kmeans.labels_
score_samples = silhouette_samples(iris.data, irisDF['cluster'])
print('silhouette_samples( ) return 값의 shape' , score_samples.shape)
irisDF['silhouette_coeff'] = score_samples
average_score = silhouette_score(iris.data, irisDF['cluster'])
print('붓꽃 데이터셋 Silhouette Analysis Score:{0:.3f}'.format(average_score))
irisDF.head(3)
irisDF.groupby('cluster')['silhouette_coeff'].mean()### 여러개의 클러스터링 갯수를 List로 입력 받아 각각의 실루엣 계수를 면적으로 시각화한 함수 작성
def visualize_silhouette(cluster_lists, X_features):
from sklearn.datasets import make_blobs
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_samples, silhouette_score
import matplotlib.pyplot as plt
import matplotlib.cm as cm
import math
n_cols = len(cluster_lists)
fig, axs = plt.subplots(figsize=(4*n_cols, 4), nrows=1, ncols=n_cols)
for ind, n_cluster in enumerate(cluster_lists):
# KMeans 클러스터링 수행하고, 실루엣 스코어와 개별 데이터의 실루엣 값 계산.
clusterer = KMeans(n_clusters = n_cluster, max_iter=500, random_state=0)
cluster_labels = clusterer.fit_predict(X_features)
sil_avg = silhouette_score(X_features, cluster_labels)
sil_values = silhouette_samples(X_features, cluster_labels)
y_lower = 10
axs[ind].set_title('Number of Cluster : '+ str(n_cluster)+'\n' \
'Silhouette Score :' + str(round(sil_avg,3)) )
axs[ind].set_xlabel("The silhouette coefficient values")
axs[ind].set_ylabel("Cluster label")
axs[ind].set_xlim([-0.1, 1])
axs[ind].set_ylim([0, len(X_features) + (n_cluster + 1) * 10])
axs[ind].set_yticks([]) # Clear the yaxis labels / ticks
axs[ind].set_xticks([0, 0.2, 0.4, 0.6, 0.8, 1])
for i in range(n_cluster):
ith_cluster_sil_values = sil_values[cluster_labels==i]
ith_cluster_sil_values.sort()
size_cluster_i = ith_cluster_sil_values.shape[0]
y_upper = y_lower + size_cluster_i
color = cm.nipy_spectral(float(i) / n_cluster)
axs[ind].fill_betweenx(np.arange(y_lower, y_upper), 0, ith_cluster_sil_values, \
facecolor=color, edgecolor=color, alpha=0.7)
axs[ind].text(-0.05, y_lower + 0.5 * size_cluster_i, str(i))
y_lower = y_upper + 10
axs[ind].axvline(x=sil_avg, color="red", linestyle="--")
from sklearn.datasets import make_blobs
X, y = make_blobs(n_samples=500, n_features=2, centers=4, cluster_std=1, \
center_box=(-10.0, 10.0), shuffle=True, random_state=1)
visualize_silhouette([ 2, 3, 4, 5], X)
from sklearn.datasets import load_iris
iris=load_iris()
visualize_silhouette([ 2, 3, 4,5 ], iris.data)CH.7.3 _평균 이동
평균 이동(Mean Shift)의 개요
- 평균 이동 : K-평균과 유사하게 중심을 군집의 중심으로 지속적으로 움직이면서 군집화를 수행.
- K-평균이 중심에 소속된 데이터의 평균 거리 중심으로 이동하는데 반해, 평균 이동은 중심을 데이터가 모여 있는 밀도가 가장 높은 곳으로 이동.
- 데이터의 분포도를 이용해 군집 중심점을 찾음 → 이를 위해 확률 밀도 함수 이용
- 가장 집중적으로 데이터가 모여있어 확률 밀도 함수가 피크인 점을 군집 중심점으로 선정, 일반적으로 주어진 모델의 확률 밀도 함수를 찾기 위해 KDE 이용
- 평균 이동 군집화 : 특정 데이터를 반경 내의 데이터 분포 확률 밀도가 가장 높은 곳으로 이동하기 위해 주변 데이터와의 거리 값을 KDE 함수 값으로 입력한 뒤, 그 반환 값을 현재 위치에서 업데이트하면서 이동하는 방식을 취함. → 이 방식을 전체 데이터에 반복적으로 적용하면서 데이터의 군집 중심점을 찾아냄.

- KDE : 관측된 데이터 각각에 커널 함수를 적용한 값을 모두 더한 뒤 데이터 건수로 나눠 확률 밀도 함수 추정
- 대표적으로 커널 함수로서 가우시안 분포 함수가 사용됨.
- 확률 밀도 함수 (PDF) : 확률 변수의 분포를 나타내는 함수. 감마 분포, 정규 분포, t-분포 등이 있음.
왼쪽은 개별 데이터에 가우시안 커널 함수 적용, 오른쪽은 적용 값을 모두 더한 KDE 결과

- KDE는 다음과 같은 함수식으로 표현 가능

K : 커널 함수, x : 확률 변수값, xi : 관측값, h : 대역폭 (meanshift에서 bandwidth와 같음)
- 대역폭 h : KDE 형태를 부드러운 형태로 평활화하는데 적용. 이 h를 어떻게 설정하느냐에 따라 확률 밀도 추정 성능을 크게 좌우할 수 있음.
- 작은 h값은 변동성이 큰 방식으로 확률 밀도 함수를 추정하기에 과적합하기 쉬움.
- 큰 h값인 경우는 지나치게 단순화된 방식으로 확률 밀도 함수를 추정하며 과소적합하기 쉬움
- → 적절한 h를 구하는 것은 KDE 기반의 평균 이동 군집화에서 매우 중요
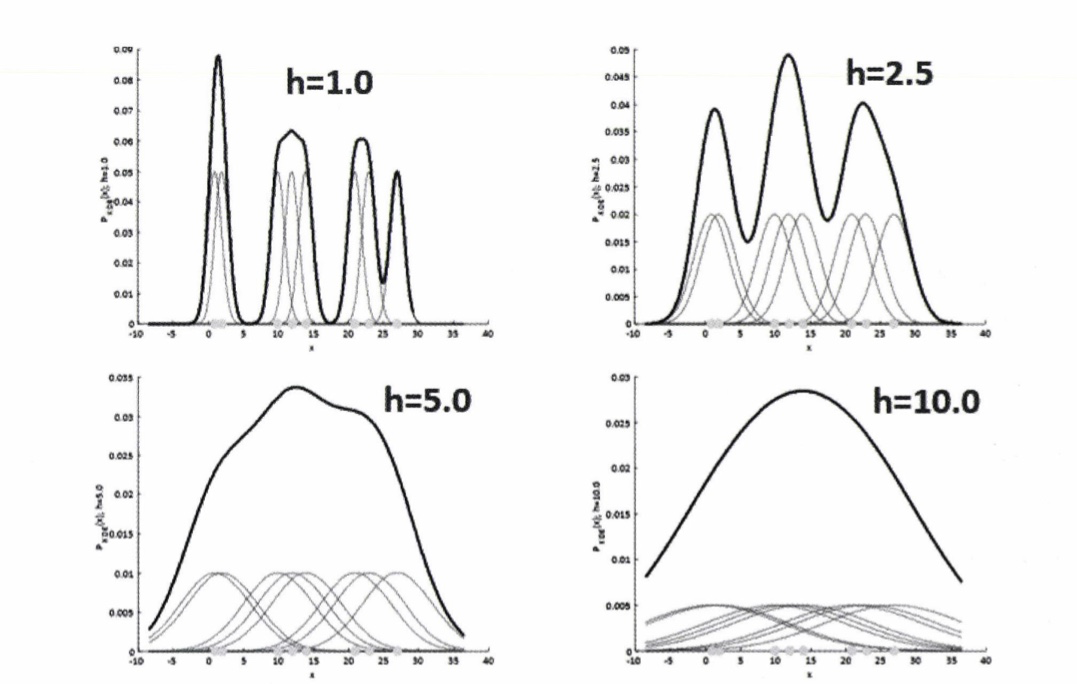
- 일반적으로 평균 이동 군집화는 대역폭이 클수록 평활화된 KDE로 인해 적은 수의 군집 중심점을 가지며, 대역폭이 적을수록 많은 수의 군집 중심점을 가짐.
→ 사이킷 런에서 최적의 대역폭 계산을 위한 estimate_bandwith() 함수를 제공
- 평균 이동의 장점
- 데이터 세트의 형태를 특정 형태로 가정, 특정 분포도 기반의 모델로 가정하지 않기 때문에 좀 더 유연한 군집화 가능
- 이상치의 영향력도 크지 않고, 미리 군집의 개수를 정할 필요 없음.
- 단점
- 알고리즘의 수행시간이 오래 걸리고, 무엇보다 bandwidth의 크기에 따른 군집화 영향도가 매우 큼
→ 분석보다는 컴퓨터 비전 영역에서 더 많이 사용
import numpy as np
from sklearn.datasets import make_blobs
from sklearn.cluster import MeanShift
X, y = make_blobs(n_samples=200, n_features=2, centers=3,
cluster_std=0.8, random_state=0)
meanshift= MeanShift(bandwidth=0.9)
cluster_labels = meanshift.fit_predict(X)
print('cluster labels 유형:', np.unique(cluster_labels))
meanshift= MeanShift(bandwidth=1)
cluster_labels = meanshift.fit_predict(X)
print('cluster labels 유형:', np.unique(cluster_labels))
from sklearn.cluster import estimate_bandwidth
bandwidth = estimate_bandwidth(X,quantile=0.2)
print('bandwidth 값:', round(bandwidth,3))
import pandas as pd
clusterDF = pd.DataFrame(data=X, columns=['ftr1', 'ftr2'])
clusterDF['target'] = y
#최적의 bandwidth 계산
best_bandwidth = estimate_bandwidth(X, quantile=0.2)
meanshift= MeanShift(bandwidth=1)
cluster_labels = meanshift.fit_predict(X)
print('cluster labels 유형:',np.unique(cluster_labels))
import matplotlib.pyplot as plt
%matplotlib inline
clusterDF['meanshift_label'] = cluster_labels
centers = meanshift.cluster_centers_
unique_labels = np.unique(cluster_labels)
markers=['o', 's', '^', 'x', '*']
for label in unique_labels:
label_cluster = clusterDF[clusterDF['meanshift_label']==label]
center_x_y = centers[label]
plt.scatter(x=label_cluster['ftr1'], y=label_cluster['ftr2'], edgecolor='k',
marker=markers[label] )
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=200, color='white',
edgecolor='k', alpha=0.9, marker=markers[label])
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=70, color='k', edgecolor='k',
marker='$%d$' % label)
plt.show()
print(clusterDF.groupby('target')['meanshift_label'].value_counts())CH.7.4 _GMM(Gaussian Mixture Model)
GMM 소개
- GMM 군집화 : 군집화를 적용하고자 하는 데이터가 여러 개의 가우시안 분포를 가진 데이터 집합들이 섞여서 생성된 것이라는 가정하에 군집화를 수행하는 방식.
- 가우시안 분포(정규 분포) : 좌우 대칭형의 종 형태를 가진 통계학에서 가장 잘 알려진 연속 확률 함수

- 정규 분포는 : 평균을 중심으로 높은 데이터 분포도를 가지고 있음., 평균이 0이고 표준편차가 1인 분포
- GMM은 데이터를 여러 개의 가우시안 분포가 섞인 것으로 간주, 섞인 데이터 분포에서 개별 유형의 가우시안 분포 추출
- GMM 설명
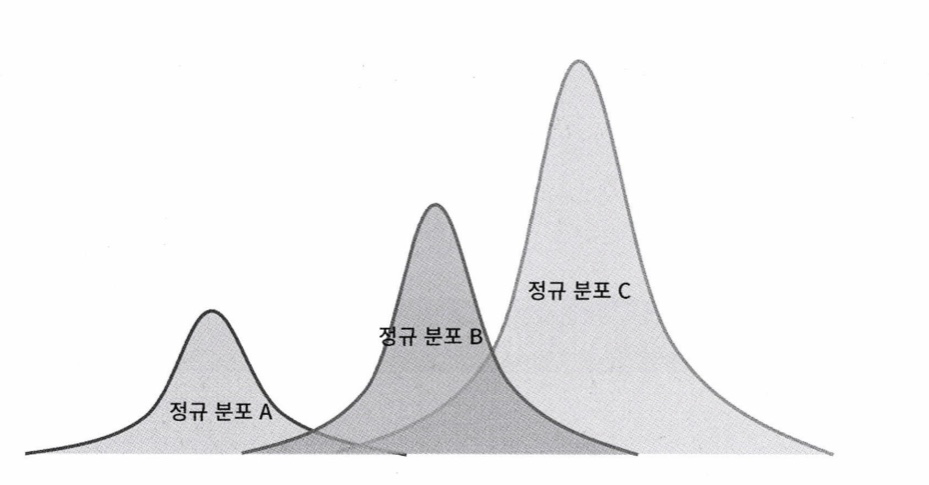
3개의 정규분포가 있다고 가정하자. 이 세 개의 정규 분포를 합치면

다음과 같이 나온다. 군집화를 수행하려는 실제 데이터 세트의 데이터 분포도가 다음과 같다면 쉽게 이 데이터 세트가 정규 분포 A,B,C가 합쳐서 된 데이터 분포도임을 알 수 있음.

전체 데이터 세트는 서로 다른 정규 분포 형태를 가진 여러 확률 분포 곡선으로 구성될 수 있으며, 이러한 서로 다른 정규 분포에 기반해 군집화를 수행하는 것이 GMM 군집화 방식
여러 개의 데이터 세트가 있다면, 이를 구성하는 여러 개의 정규 분포 곡선을 추출하고, 개별 데이터가 이 중 어떤 정규분포에 속하는지 결정하는 방식

- 이를 모수 추정이라 함. 모수 추정은 2가지를 추정하는 것
- 개별 정규 분포의 평균과 분산
- 각 데이터가 어떤 정규 분포에 해당되는지 확률
→ 사이킷 런에서는 이러한 GMM의 EM 방식을 통한 모수 추정 군집화를 위해 GaussianMixture 지원
GMM과 K-평균 비교
- KMeans : 데이터 세트가 원형의 범위를 가질수록 효율이 높아짐. 반대로 데이터가 길쭉한 타원형으로 늘어선 경우에는 군집화를 잘 수행하지 못함.
- GMM : 군집의 중심 좌표를 구할 수 없음. 다만, 유연하게 다양한 데이터 세트에 잘 적용할 수 있음.
from sklearn.datasets import load_iris
from sklearn.cluster import KMeans
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
%matplotlib inline
iris = load_iris()
feature_names = ['sepal_length','sepal_width','petal_length','petal_width']
irisDF = pd.DataFrame(data=iris.data, columns=feature_names)
irisDF['target'] = iris.target
from sklearn.mixture import GaussianMixture
gmm = GaussianMixture(n_components=3, random_state=0).fit(iris.data)
gmm_cluster_labels = gmm.predict(iris.data)
irisDF['gmm_cluster'] = gmm_cluster_labels
irisDF['target'] = iris.target
iris_result = irisDF.groupby(['target'])['gmm_cluster'].value_counts()
print(iris_result)
kmeans = KMeans(n_clusters=3, init='k-means++', max_iter=300,random_state=0).fit(iris.data)
kmeans_cluster_labels = kmeans.predict(iris.data)
irisDF['kmeans_cluster'] = kmeans_cluster_labels
iris_result = irisDF.groupby(['target'])['kmeans_cluster'].value_counts()
print(iris_result)### 클러스터링 결과를 시각화 함수
def visualize_cluster_plot(clusterobj, dataframe, label_name, iscenter=True):
if iscenter :
centers = clusterobj.cluster_centers_
unique_labels = np.unique(dataframe[label_name].values)
markers=['o', 's', '^', 'x', '*']
isNoise=False
for label in unique_labels:
label_cluster = dataframe[dataframe[label_name]==label]
if label == -1:
cluster_legend = 'Noise'
isNoise=True
else :
cluster_legend = 'Cluster '+str(label)
plt.scatter(x=label_cluster['ftr1'], y=label_cluster['ftr2'], s=70,\
edgecolor='k', marker=markers[label], label=cluster_legend)
if iscenter:
center_x_y = centers[label]
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=250, color='white',
alpha=0.9, edgecolor='k', marker=markers[label])
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=70, color='k',\
edgecolor='k', marker='$%d$' % label)
if isNoise:
legend_loc='upper center'
else: legend_loc='upper right'
plt.legend(loc=legend_loc)
plt.show()
from sklearn.datasets import make_blobs
X, y = make_blobs(n_samples=300, n_features=2, centers=3, cluster_std=0.5, random_state=0)
transformation = [[0.60834549, -0.63667341], [-0.40887718, 0.85253229]]
X_aniso = np.dot(X, transformation)
clusterDF = pd.DataFrame(data=X_aniso, columns=['ftr1', 'ftr2'])
clusterDF['target'] = y
visualize_cluster_plot(None, clusterDF, 'target', iscenter=False)
# 3개의 Cluster 기반 Kmeans 를 X_aniso 데이터 셋에 적용
kmeans = KMeans(3, random_state=0)
kmeans_label = kmeans.fit_predict(X_aniso)
clusterDF['kmeans_label'] = kmeans_label
visualize_cluster_plot(kmeans, clusterDF, 'kmeans_label',iscenter=True)
# 3개의 n_components기반 GMM을 X_aniso 데이터 셋에 적용
gmm = GaussianMixture(n_components=3, random_state=0)
gmm_label = gmm.fit(X_aniso).predict(X_aniso)
clusterDF['gmm_label'] = gmm_label
visualize_cluster_plot(gmm, clusterDF, 'gmm_label',iscenter=False)
print('### KMeans Clustering ###')
print(clusterDF.groupby('target')['kmeans_label'].value_counts())
print('\n### Gaussian Mixture Clustering ###')
print(clusterDF.groupby('target')['gmm_label'].value_counts())CH.7.5 _ DBSCAN
DBSCAN 개요
- DBSCAN : 밀도 기반 군집화의 대표 알고리즘, 간단하고 직관적인 알고리즘으로 돼있음에도 데이터의 분포가 기하학적으로 복잡한 데이터 세트에도 효과적인 군집화가 가능
- 특정 공간 내에 데이터 밀도 차이를 기반 알고리즘으로 하고 있어서 복잡한 기하학적 분포도를 가진 데이터 세트에 대해서도 군집화 수행
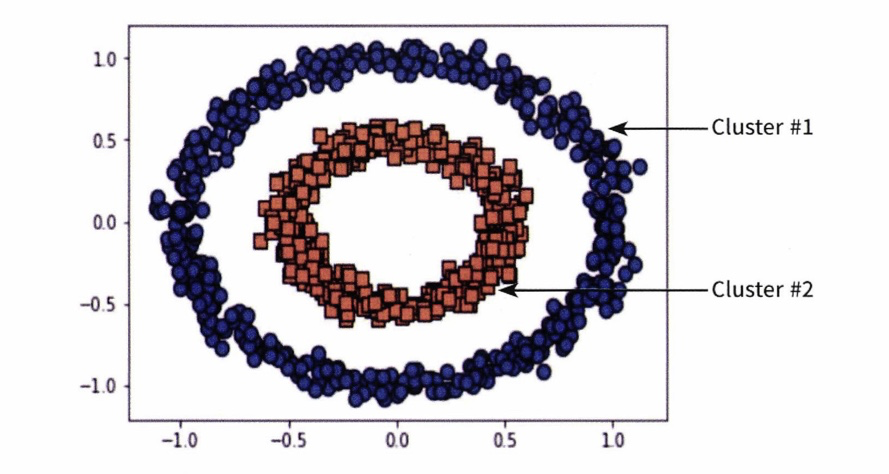
- 주요 파라미터
- 입실론 주변 영역 (epsilon) : 개별 데이터를 중심으로 입실론 반경을 가지는 원형의 영역
- 최소 데이터 개수 : 개별 데이터의 입실론 주변 영역에 포함되는 타 데이터 개수
- 입실론 데이터 포인트 정의 (주변 영역 내에 포함되는 최소 데이터 개수를 충족시키는가에 따라)
- 핵심 포인트 (core point) : 주변 영역 내에 최소 데이터 개수 이상의 타 데이터를 가지고 있을 경우 해당 데이터
- 이웃 포인트 (neighbor point) : 주변 영역 내에 위치한 타 데이터
- 경계 포인트 (border point) : 주변 영역 내 최소 데이터 개수 이상의 이웃 포인트를 가지고 있지 않지만, 핵심 포인트를 이웃 포인트로 가지고 있는 데이터
- 잡음 포인트 (noise point) : 최소 데이터 개수 이상의 이웃 포인트를 가지고 있지 않으며, 핵심 포인트도 이웃 포인트로 가지고 있지 않은 데이터

- 사이킷 런의 DBSCAN 알고리즘
- eps : 입실론 주변 영역의 반경
- min_samples : 핵심 포인트가 되기 위해 입실론 주변 영역 내에 포함돼야 할 최소 데이터 개수 (자신의 데이터 포함)
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
%matplotlib inline
iris = load_iris()
feature_names = ['sepal_length','sepal_width','petal_length','petal_width']
irisDF = pd.DataFrame(data=iris.data, columns=feature_names)
irisDF['target'] = iris.target
from sklearn.cluster import DBSCAN
dbscan = DBSCAN(eps=0.6, min_samples=8, metric='euclidean')
dbscan_labels = dbscan.fit_predict(iris.data)
irisDF['dbscan_cluster'] = dbscan_labels
irisDF['target'] = iris.target
iris_result = irisDF.groupby(['target'])['dbscan_cluster'].value_counts()
print(iris_result)
### 클러스터 결과를 클러스터링 결과 시각화 함수
def visualize_cluster_plot(clusterobj, dataframe, label_name, iscenter=True):
if iscenter :
centers = clusterobj.cluster_centers_
unique_labels = np.unique(dataframe[label_name].values)
markers=['o', 's', '^', 'x', '*']
isNoise=False
for label in unique_labels:
label_cluster = dataframe[dataframe[label_name]==label]
if label == -1:
cluster_legend = 'Noise'
isNoise=True
else :
cluster_legend = 'Cluster '+str(label)
plt.scatter(x=label_cluster['ftr1'], y=label_cluster['ftr2'], s=70,\
edgecolor='k', marker=markers[label], label=cluster_legend)
if iscenter:
center_x_y = centers[label]
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=250, color='white',
alpha=0.9, edgecolor='k', marker=markers[label])
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=70, color='k',\
edgecolor='k', marker='$%d$' % label)
if isNoise:
legend_loc='upper center'
else: legend_loc='upper right'
plt.legend(loc=legend_loc)
plt.show()
from sklearn.decomposition import PCA
# 2차원으로 시각화하기 위해 PCA n_componets=2로 피처 데이터 세트 변환
pca = PCA(n_components=2, random_state=0)
pca_transformed = pca.fit_transform(iris.data)
# visualize_cluster_2d( ) 함수는 ftr1, ftr2 컬럼을 좌표에 표현하므로 PCA 변환값을 해당 컬럼으로 생성
irisDF['ftr1'] = pca_transformed[:,0]
irisDF['ftr2'] = pca_transformed[:,1]
visualize_cluster_plot(dbscan, irisDF, 'dbscan_cluster', iscenter=False)
visualize_cluster_plot(dbscan, irisDF, 'dbscan_cluster', iscenter=False)
from sklearn.cluster import DBSCAN
dbscan = DBSCAN(eps=0.8, min_samples=8, metric='euclidean')
dbscan_labels = dbscan.fit_predict(iris.data)
irisDF['dbscan_cluster'] = dbscan_labels
irisDF['target'] = iris.target
iris_result = irisDF.groupby(['target'])['dbscan_cluster'].value_counts()
print(iris_result)
visualize_cluster_plot(dbscan, irisDF, 'dbscan_cluster', iscenter=False)
dbscan = DBSCAN(eps=0.6, min_samples=16, metric='euclidean')
dbscan_labels = dbscan.fit_predict(iris.data)
irisDF['dbscan_cluster'] = dbscan_labels
irisDF['target'] = iris.target
iris_result = irisDF.groupby(['target'])['dbscan_cluster'].value_counts()
print(iris_result)
visualize_cluster_plot(dbscan, irisDF, 'dbscan_cluster', iscenter=False)
from sklearn.datasets import make_circles
X, y = make_circles(n_samples=1000, shuffle=True, noise=0.05, random_state=0, factor=0.5)
clusterDF = pd.DataFrame(data=X, columns=['ftr1', 'ftr2'])
clusterDF['target'] = y
visualize_cluster_plot(None, clusterDF, 'target', iscenter=False)
# KMeans로 make_circles( ) 데이터 셋을 클러스터링 수행.
from sklearn.cluster import KMeans
kmeans = KMeans(n_clusters=2, max_iter=1000, random_state=0)
kmeans_labels = kmeans.fit_predict(X)
clusterDF['kmeans_cluster'] = kmeans_labels
visualize_cluster_plot(kmeans, clusterDF, 'kmeans_cluster', iscenter=True)
# GMM으로 make_circles( ) 데이터 셋을 클러스터링 수행.
from sklearn.mixture import GaussianMixture
gmm = GaussianMixture(n_components=2, random_state=0)
gmm_label = gmm.fit(X).predict(X)
clusterDF['gmm_cluster'] = gmm_label
visualize_cluster_plot(gmm, clusterDF, 'gmm_cluster', iscenter=False)
# DBSCAN으로 make_circles( ) 데이터 셋을 클러스터링 수행.
from sklearn.cluster import DBSCAN
dbscan = DBSCAN(eps=0.2, min_samples=10, metric='euclidean')
dbscan_labels = dbscan.fit_predict(X)
clusterDF['dbscan_cluster'] = dbscan_labels
visualize_cluster_plot(dbscan, clusterDF, 'dbscan_cluster', iscenter=False)
CH.7.6 _ 실습
import pandas as pd
import datetime
import math
import numpy as np
import matplotlib.pyplot as plt
%matplotlib inline
retail_df = pd.read_excel(io='Online Retail.xlsx')
retail_df.info()
retail_df = retail_df[retail_df['Quantity'] > 0]
retail_df = retail_df[retail_df['UnitPrice'] > 0]
retail_df = retail_df[retail_df['CustomerID'].notnull()]
print(retail_df.shape)
retail_df.isnull().sum()
retail_df['Country'].value_counts()[:5]
retail_df = retail_df[retail_df['Country']=='United Kingdom']
print(retail_df.shape)
##RFM 기반 데이터 가공
retail_df['sale_amount'] = retail_df['Quantity'] * retail_df['UnitPrice']
retail_df['CustomerID'] = retail_df['CustomerID'].astype(int)
retail_df.groupby(['InvoiceNo','StockCode'])['InvoiceNo'].count().mean()
# Recency-- InvoiceDate 컬럼의 max() 에서 데이터 가공
# Frequency-- InvoiceNo 컬럼의 count() , Monetary value는 sale_amount 컬럼의 sum()
aggregations = {
'InvoiceDate': 'max',
'InvoiceNo': 'count',
'sale_amount':'sum'
}
cust_df = retail_df.groupby('CustomerID').agg(aggregations)
# groupby된 결과 컬럼값을 Recency, Frequency, Monetary로 변경
cust_df = cust_df.rename(columns = {'InvoiceDate':'Recency',
'InvoiceNo':'Frequency',
'sale_amount':'Monetary'
}
)
cust_df = cust_df.reset_index()
import datetime as dt
cust_df['Recency'] = dt.datetime(2011,12,10) - cust_df['Recency']
cust_df['Recency'] = cust_df['Recency'].apply(lambda x: x.days+1)
print('cust_df 로우와 컬럼 건수는 ',cust_df.shape)
cust_df.head(3)
## RFM 기반 고객 세그먼테이션
fig, (ax1,ax2,ax3) = plt.subplots(figsize=(12,4), nrows=1, ncols=3)
ax1.set_title('Recency Histogram')
ax1.hist(cust_df['Recency'])
ax2.set_title('Frequency Histogram')
ax2.hist(cust_df['Frequency'])
ax3.set_title('Monetary Histogram')
ax3.hist(cust_df['Monetary'])
cust_df[['Recency','Frequency','Monetary']].describe()
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_score, silhouette_samples
X_features = cust_df[['Recency','Frequency','Monetary']].values
X_features_scaled = StandardScaler().fit_transform(X_features)
kmeans = KMeans(n_clusters=3, random_state=0)
labels = kmeans.fit_predict(X_features_scaled)
cust_df['cluster_label'] = labels
print('실루엣 스코어는 : {0:.3f}'.format(silhouette_score(X_features_scaled,labels)))### 여러개의 클러스터링 갯수를 List로 입력 받아 각각의 실루엣 계수를 면적으로 시각화한 함수 작성
def visualize_silhouette(cluster_lists, X_features):
from sklearn.datasets import make_blobs
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_samples, silhouette_score
import matplotlib.pyplot as plt
import matplotlib.cm as cm
import math
n_cols = len(cluster_lists)
fig, axs = plt.subplots(figsize=(4*n_cols, 4), nrows=1, ncols=n_cols)
for ind, n_cluster in enumerate(cluster_lists):
clusterer = KMeans(n_clusters = n_cluster, max_iter=500, random_state=0)
cluster_labels = clusterer.fit_predict(X_features)
sil_avg = silhouette_score(X_features, cluster_labels)
sil_values = silhouette_samples(X_features, cluster_labels)
y_lower = 10
axs[ind].set_title('Number of Cluster : '+ str(n_cluster)+'\n' \
'Silhouette Score :' + str(round(sil_avg,3)) )
axs[ind].set_xlabel("The silhouette coefficient values")
axs[ind].set_ylabel("Cluster label")
axs[ind].set_xlim([-0.1, 1])
axs[ind].set_ylim([0, len(X_features) + (n_cluster + 1) * 10])
axs[ind].set_yticks([]) # Clear the yaxis labels / ticks
axs[ind].set_xticks([0, 0.2, 0.4, 0.6, 0.8, 1])
for i in range(n_cluster):
ith_cluster_sil_values = sil_values[cluster_labels==i]
ith_cluster_sil_values.sort()
size_cluster_i = ith_cluster_sil_values.shape[0]
y_upper = y_lower + size_cluster_i
color = cm.nipy_spectral(float(i) / n_cluster)
axs[ind].fill_betweenx(np.arange(y_lower, y_upper), 0, ith_cluster_sil_values, \
facecolor=color, edgecolor=color, alpha=0.7)
axs[ind].text(-0.05, y_lower + 0.5 * size_cluster_i, str(i))
y_lower = y_upper + 10
axs[ind].axvline(x=sil_avg, color="red", linestyle="--")
### 여러개의 클러스터링 갯수를 List로 입력 받아 각각의 클러스터링 결과를 시각화
def visualize_kmeans_plot_multi(cluster_lists, X_features):
from sklearn.cluster import KMeans
from sklearn.decomposition import PCA
import pandas as pd
import numpy as np
n_cols = len(cluster_lists)
fig, axs = plt.subplots(figsize=(4*n_cols, 4), nrows=1, ncols=n_cols)
# 입력 데이터의 FEATURE가 여러개일 경우 2차원 데이터 시각화가 어려우므로 PCA 변환하여 2차원 시각화
pca = PCA(n_components=2)
pca_transformed = pca.fit_transform(X_features)
dataframe = pd.DataFrame(pca_transformed, columns=['PCA1','PCA2'])
# 리스트에 기재된 클러스터링 갯수들을 차례로 iteration 수행하면서 KMeans 클러스터링 수행하고 시각화
for ind, n_cluster in enumerate(cluster_lists):
clusterer = KMeans(n_clusters = n_cluster, max_iter=500, random_state=0)
cluster_labels = clusterer.fit_predict(pca_transformed)
dataframe['cluster']=cluster_labels
unique_labels = np.unique(clusterer.labels_)
markers=['o', 's', '^', 'x', '*']
for label in unique_labels:
label_df = dataframe[dataframe['cluster']==label]
if label == -1:
cluster_legend = 'Noise'
else :
cluster_legend = 'Cluster '+str(label)
axs[ind].scatter(x=label_df['PCA1'], y=label_df['PCA2'], s=70,\
edgecolor='k', marker=markers[label], label=cluster_legend)
axs[ind].set_title('Number of Cluster : '+ str(n_cluster))
axs[ind].legend(loc='upper right')
plt.show()
visualize_silhouette([2,3,4,5],X_features_scaled)
visualize_kmeans_plot_multi([2,3,4,5],X_features_scaled)

### Log 변환을 통해 데이터 변환
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_score, silhouette_samples
cust_df['Recency_log'] = np.log1p(cust_df['Recency'])
cust_df['Frequency_log'] = np.log1p(cust_df['Frequency'])
cust_df['Monetary_log'] = np.log1p(cust_df['Monetary'])
X_features = cust_df[['Recency_log','Frequency_log','Monetary_log']].values
X_features_scaled = StandardScaler().fit_transform(X_features)
kmeans = KMeans(n_clusters=3, random_state=0)
labels = kmeans.fit_predict(X_features_scaled)
cust_df['cluster_label'] = labels
print('실루엣 스코어는 : {0:.3f}'.format(silhouette_score(X_features_scaled,labels)))
visualize_silhouette([2,3,4,5],X_features_scaled)
visualize_kmeans_plot_multi([2,3,4,5],X_features_scaled)

CH.7.7 _ 정리
K-평균 : 거리 기반으로 군집 중심점을 이동시키면서 군집화 수행
--> 쉽고 직관적이지만 복잡한 구조 데이터셋에서는 적용 어려움 & 군집 개수 최적화 어려움
--> 군집 평가를 위해 실루엣 계수 이용
평균 이동 : 거리 중심이 아닌 데이터가 모여 있는 밀도가 높은 쪽으로 군집 중심점 이동시키며 수행
GMM : 전체 데이터 셋에서 서로 다른 정규 분포 형태를 추출해 다른 정규 부포를 가진 데이터 셋을 각각 군집화 하는 것
DBSCAN : 밀도 기반의 군집화 - 입실론 주변 영역의 최소 데이터 개수 충족 여부 파악 --> 직접 접근 가능한 핵심 포인트를 연결하며 군집화 진행
'Self-Taught > Machine Learning' 카테고리의 다른 글
| ECC 프로젝트) 1차 회의용 _ 데이터 분석 내용 (0) | 2025.01.22 |
|---|---|
| 머신러닝 완벽 가이드 _ CH.8 : 텍스트 분석 (2) (2) | 2024.12.26 |
| 파이썬 머신러닝 완벽 가이드 _CH.6 (1) | 2024.11.23 |
| 파이썬 머신러닝 완벽 가이드 _CH.5 (0) | 2024.11.16 |
| 파이썬 머신러닝 완벽 가이드 _ CH.4.7~4.12 (1) | 2024.11.09 |



